Apprenez à utiliser les outils OCR, Apache Spark et d'autres composants Apache Hadoop pour traiter des images PDF à grande échelle.
Les technologies de reconnaissance optique de caractères (OCR) ont considérablement progressé au cours des 20 dernières années. Cependant, pendant cette période, peu ou pas d'efforts ont été déployés pour associer l'OCR à des architectures distribuées telles qu'Apache Hadoop afin de traiter un grand nombre d'images en temps quasi réel.
Dans cet article, vous apprendrez à utiliser des outils open source standard avec des composants Hadoop tels qu'Apache Spark, Apache Solr et Apache HBase pour faire exactement cela pour un cas d'utilisation d'informations sur un dispositif médical. Plus précisément, vous utiliserez un ensemble de données public pour convertir le texte narratif en champs interrogeables.
Bien que cet exemple se concentre sur les informations sur les dispositifs médicaux, il peut être appliqué dans de nombreux autres scénarios où le traitement et la persistance des images sont nécessaires. Les compagnies d'assurance, par exemple, peuvent rendre tous leurs documents numérisés dans les dossiers de réclamation consultables pour une meilleure résolution des réclamations. De même, le service de la chaîne d'approvisionnement d'une usine de fabrication pourrait numériser toutes les fiches techniques des fournisseurs de pièces et les rendre consultables par les analystes.
Cas d'utilisation :enregistrement des dispositifs médicaux
Ces dernières années ont vu une vague de changements dans le domaine de l'enregistrement électronique des médicaments. La norme ISO IDMP (identification des produits médicaux) est l'un de ces formats de message pour l'enregistrement des produits et des substances qu'ils contiennent, l'ID du médicament, l'ID de l'emballage et l'ID du lot étant utilisés pour suivre les produits en cas d'expériences indésirables, d'actes illégaux. l'importation, la contrefaçon et d'autres problèmes de pharmacovigilance. La norme demande que non seulement les nouveaux produits doivent être enregistrés, mais que le dossier plus ancien/archivé de chaque produit auquel le public pourrait être exposé doit également être fourni sous forme électronique.
Pour se conformer aux normes IDMP dans différentes entreprises, les entreprises doivent être en mesure d'extraire et de traiter des données provenant de plusieurs sources de données, telles que RDBMS ainsi que, dans certains cas, des fiches techniques de produits héritées. S'il est bien connu d'ingérer des données de RDBMS via des technologies comme Apache Sqoop, le traitement de documents hérités nécessite un peu plus de travail. Pour la plupart, les documents doivent être ingérés et le texte pertinent doit être extrait par programme à grande échelle à l'aide des technologies OCR existantes.
Ensemble de données
Nous utiliserons un ensemble de données de la FDA qui contient tous les dossiers 510(k) jamais soumis par les fabricants de dispositifs médicaux depuis 1976. La section 510(k) de la Food, Drug and Cosmetic Act exige que les fabricants de dispositifs qui doivent s'enregistrer, notifient FDA de son intention de commercialiser un dispositif médical au moins 90 jours à l'avance.
Cet ensemble de données est utile pour plusieurs raisons dans ce cas :
- Les données sont gratuites et dans le domaine public.
- Les données s'inscrivent parfaitement dans la réglementation européenne, qui entre en vigueur en juillet 2016 (où les fabricants doivent se conformer aux nouvelles normes de données). Les remplissages de la FDA contiennent des informations importantes permettant d'obtenir une vue complète de l'IDMP.
- Le format des documents (PDF) nous permet de démontrer des techniques OCR simples mais efficaces lorsque nous traitons des documents de plusieurs formats.
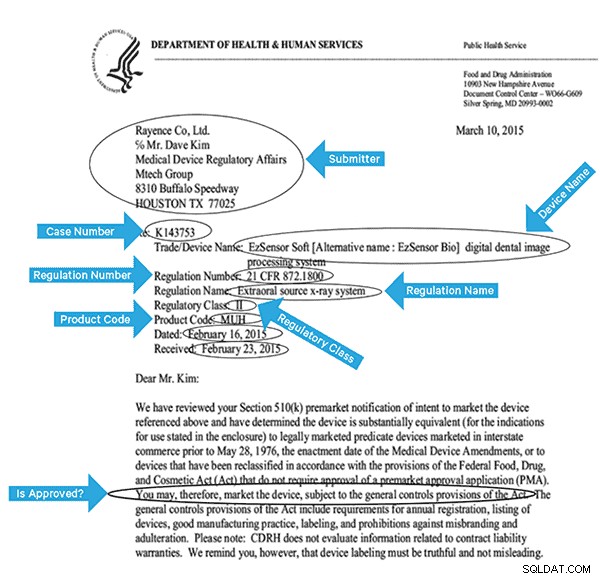
Pour indexer efficacement ces données, nous devrons extraire certains champs des images. Vous trouverez ci-dessous un exemple de document, avec les champs potentiels pouvant être extraits.
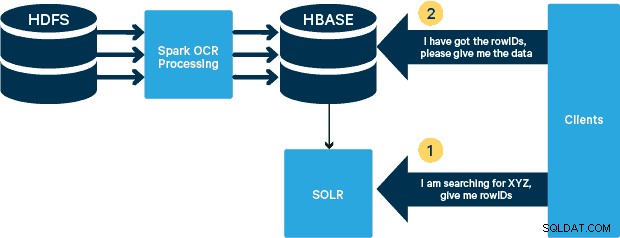
Architecture de haut niveau
Pour ce cas d'utilisation, les fichiers PDF sont stockés dans HDFS et traités à l'aide des bibliothèques Spark et OCR. (L'étape d'ingestion sort du cadre de cet article, mais elle pourrait être aussi simple que d'exécuter hdfs -dfs -put ou en utilisant une interface webhdfs.) Spark permet l'utilisation d'un code presque identique dans une application Spark Streaming pour un streaming en temps quasi réel, et HBase est un support de stockage parfait pour un accès aléatoire à faible latence et est bien adapté pour stocker des images, avec la nouvelle fonctionnalité MOB, pour démarrer. Cloudera Search (qui repose sur Apache Solr) est la seule solution de recherche qui s'intègre nativement à HBase, vous permettant ainsi de créer des index secondaires.
Configuration de la table des dispositifs médicaux dans HBase
Nous garderons le schéma de notre cas d'utilisation simple. Le rowID sera le nom du fichier, et il y aura deux familles de colonnes :« info » et « obj ». La famille de colonnes "info" contiendra tous les champs que nous avons extraits des images. La famille de colonnes "obj" contiendra les octets de l'objet binaire réel, dans ce cas PDF. Le nom de la table dans notre cas sera "mdds".
Nous tirerons parti de la fonctionnalité HBase MOB (objet moyen) introduite dans HBASE-11339. Pour configurer HBase pour gérer MOB, quelques étapes supplémentaires sont nécessaires, mais, pour plus de commodité, des instructions peuvent être trouvées sur ce lien.
Il existe de nombreuses façons de créer la table dans HBase par programme (API Java, API REST ou une méthode similaire). Ici, nous allons utiliser le shell HBase pour créer la table "mdds" (en utilisant intentionnellement un nom de famille de colonne descriptif pour faciliter le suivi). Nous voulons que la famille de colonnes "info" soit répliquée dans Solr, mais pas les données MOB.
La commande ci-dessous créera la table et activera la réplication sur une famille de colonnes appelée "info". Il est crucial de spécifier l'option REPLICATION_SCOPE => '1' , sinon l'indexeur HBase Lily ne recevra aucune mise à jour de HBase. Nous voulons utiliser le chemin MOB dans HBase pour les objets de plus de 10 Mo. Pour ce faire, nous créons également une autre famille de colonnes, appelée "obj", en utilisant les paramètres suivants pour les MOB :
IS_MOB => vrai, MOB_THRESHOLD => 10240000
Le IS_MOB Le paramètre spécifie si cette famille de colonnes peut stocker des MOB, tandis que MOB_THRESHOLD spécifie après quelle taille l'objet doit être pour qu'il soit considéré comme un MOB. Alors, créons le tableau :
create 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
Pour confirmer que la table a été créée correctement, exécutez la commande suivante dans le shell HBase :
hbase(main):001:0> describe 'mdds'Table mdds is ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1' , VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'POUR TOUJOURS', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}2 ligne(s) en 0.3440 secondes Traitement des images numérisées avec Tesseract
L'OCR a parcouru un long chemin en termes de gestion des variations de police, du bruit d'image et des problèmes d'alignement. Ici, nous utiliserons le moteur OCR open source Tesseract, qui a été initialement développé en tant que logiciel propriétaire dans les laboratoires HP. Le développement de Tesseract est depuis sorti en tant que logiciel open source et sponsorisé par Google depuis 2006.
Tesseract est une bibliothèque de logiciels hautement portable. Il utilise la bibliothèque de traitement d'image Leptonica pour générer une image binaire en effectuant un seuillage adaptatif sur une image grise ou colorée.
Le traitement suit un pipeline traditionnel étape par étape. Voici le déroulement approximatif des étapes :
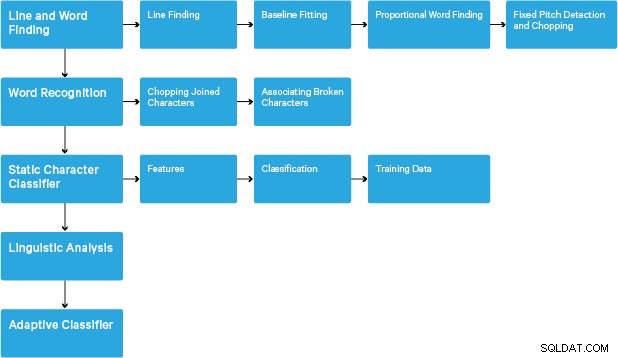
Le traitement commence par une analyse des composants connectés, qui se traduit par le stockage des composants trouvés. Cette étape permet d'inspecter l'imbrication des contours et le nombre de contours enfants et petits-enfants.
À ce stade, les contours sont rassemblés, uniquement par imbrication, dans des objets binaires volumineux (BLOB). Les BLOB sont organisés en lignes de texte, et les lignes et les régions sont analysées pour un pas fixe ou un texte proportionnel. Les lignes de texte sont divisées en mots différemment selon le type d'espacement des caractères. Le texte à espacement fixe est coupé immédiatement par les cellules de caractère. Le texte proportionnel est divisé en mots à l'aide d'espaces définis et d'espaces flous.
La reconnaissance se déroule alors comme un processus en deux étapes. Dans le premier passage, une tentative est faite pour reconnaître chaque mot à tour de rôle. Chaque mot satisfaisant est transmis à un classificateur adaptatif en tant que données d'apprentissage. Le classificateur adaptatif a alors la possibilité de reconnaître plus précisément le texte plus bas sur la page. Étant donné que le classificateur adaptatif a peut-être appris quelque chose d'utile trop tard pour apporter une contribution vers le haut de la page, une seconde passe est exécutée sur la page, dans laquelle les mots qui n'ont pas été suffisamment bien reconnus sont à nouveau reconnus. Une phase finale résout les espaces flous et vérifie des hypothèses alternatives pour la hauteur x afin de localiser le texte en petites majuscules.
Tesseract dans sa forme actuelle est entièrement compatible Unicode et formé pour plusieurs langues. D'après nos recherches, il s'agit de l'une des bibliothèques open source les plus précises disponibles pour l'OCR. Comme mentionné précédemment, Tesseract utilise Leptonica. Nous utilisons également Ghostscript pour diviser les fichiers PDF en images. (Vous pouvez diviser le format de compression d'image de votre choix; nous avons choisi PNG.) Ces trois bibliothèques sont écrites en C++, et pour les invoquer à partir de programmes Java/Scala, nous devons utiliser des implémentations des interfaces natives Java correspondantes. Dans notre travail, nous utilisons les liaisons JNI de JavaPresets. (Les instructions de construction se trouvent ci-dessous.) Nous avons utilisé Scala pour écrire le pilote Spark.
val renderer :SimpleRenderer =nouveau SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica lit les images fractionnées de l'étape précédente.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix :PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity( ))
Nous utilisons ensuite les appels de l'API Tesseract pour extraire le texte. Nous supposons que les documents sont en anglais ici, donc le deuxième paramètre de la méthode Init est "eng".
val api :TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Une fois les images traitées, nous extrayons certains champs du texte et les envoyons à HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unit ={ /** Configurer et ouvrir une connexion HBase */ val mddsTbl =_conn.getTable( TableName. valueOf( "mdds" )); val cf ="info" val put =new Put( Bytes.toBytes( fileName )) /** * Extrayez les champs ici à l'aide de Regexes * Créez des objets Put et envoyez-les à HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe :(\w\d\d\d\d\d\d).*"" ".r …….. lignes correspondent { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( addr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "ne correspond pas à une expression régulière" ) } ……. lines.split("\n").foreach { val regNumRegex ="""Numéro de la réglementation :\s+(.+)""".r val regNameRegex ="""Nom de la réglementation :\s+(.+)""" .r …….. ……. _ match { case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "texte"), Bytes.toBytes( lines )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( put ) …….}
Si vous examinez attentivement le code ci-dessus, juste avant d'envoyer l'objet Put à HBase, nous insérons les octets PDF bruts dans la famille de colonnes "obj" de la table. Nous utilisons HBase comme couche de stockage pour les champs extraits ainsi que l'image brute. Cela permet à l'application d'extraire rapidement et facilement l'image d'origine, si nécessaire. Le code complet peut être trouvé ici. (Il convient de noter que même si nous avons utilisé des API HBase standard pour créer des objets Put pour HBase, dans un système de production réel, il serait judicieux d'envisager d'utiliser des API SparkOnHBase, qui permettent des mises à jour par lots de HBase à partir de Spark RDD.)
Pipeline d'exécution
Nous avons pu traiter chaque PDF dans un cadre en série. Pour dimensionner le traitement, nous avons choisi de traiter ces PDF de manière distribuée à l'aide de Spark. Le tableau suivant montre comment nous combinons différentes étapes de ce traitement pour transformer le flux de travail en un simple appel de macro à partir de Spark et charger les données dans HBase.
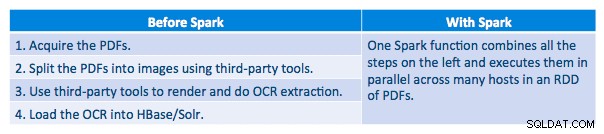
Nous avons également essayé de faire une comparaison entre les méthodes de sérialisation, mais, avec notre ensemble de données, nous n'avons pas constaté de différence significative de performances.
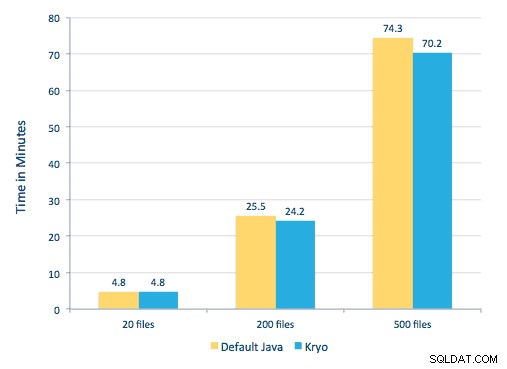
Configuration de l'environnement
Matériel utilisé :cluster à cinq nœuds avec 15 Go de mémoire, 4 processeurs virtuels et 2 disques SSD de 40 Go
Puisque nous utilisions des bibliothèques C++ pour le traitement, nous avons utilisé les liaisons JNI qui peuvent être trouvées ici.
Créez les liaisons JNI pour Tesseract et Leptonica à partir des préréglages javaCPP :
-
- Sur tous les nœuds :
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel
git clone https://github.com/bytedeco/javacpp-presets.git cd javacpp-presets - Construire Leptonica.
cd leptonica./cppbuild.sh install leptonicacd cppbuild/linux-x86_64/leptonica-1.72/LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&sudo make installcd ../../../mvn clean installcd ..
- Construisez Tesseract.
cd tesseract./cppbuild.sh install tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- Créer des préréglages javaCPP.
mvn clean install --projects leptonica,tesseract
Nous utilisons Ghostscript pour extraire les images des PDF. Les instructions pour construire Ghostscript, correspondant aux versions de Tesseract et Leptonica utilisées ici, sont les suivantes. (Assurez-vous que Ghostscript n'est pas installé dans le système via le gestionnaire de packages.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo make &&make soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps(Selon votre ldpath paramètre, vous devrez peut-être le faire) :sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Assurez-vous que toutes les bibliothèques nécessaires se trouvent dans le chemin de classe. Nous plaçons tous les fichiers jar pertinents dans un répertoire appelé lib. La virgule est importante ci-dessous :
$ pour je dans `ls lib/*` ; exportez MY_JARS=./$i,$MY_JARS ; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Nous invoquons le programme Spark comme suit. Nous devons spécifier extraLibraryPath pour les bibliothèques Ghostscript natives ; l'autre conf est nécessaire pour Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Création d'une collection Solr
Solr s'intègre de manière assez transparente à HBase via Lily HBase Indexer. Pour comprendre comment s'effectue l'intégration de Lily Indexer avec HBase, vous pouvez consulter notre article précédent dans la section "Comprendre HBase Replication et Lily HBase Indexer".
Ci-dessous, nous décrivons les étapes à suivre pour créer les index :
- Générez un exemple de fichier de configuration schema.xml :
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg
- Modifier le fichier schema.xml dans
$HOME/solrcfg , en spécifiant les champs dont nous avons besoin pour notre collection. Le fichier complet est disponible ici.
- Téléchargez les configurations Solr sur ZooKeeper :
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg
- Générez la collection Solr avec 2 partitions (-s 2) et 2 répliques (-r 2) :
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
Dans la commande ci-dessus, nous avons créé une collection Solr avec deux paramètres de fragments (-s 2) et deux répliques (-r 2). Les paramètres étaient suffisants pour notre corpus, mais dans un déploiement réel, il faudrait définir le nombre en fonction d'autres considérations en dehors de notre champ de discussion ici.
Enregistrement de l'indexeur
Cette étape est nécessaire pour ajouter et configurer l'indexeur et la réplication HBase. La commande ci-dessous mettra à jour ZooKeeper et ajoutera mdds_indexer en tant qu'homologue de réplication pour HBase. Il insérera également des configurations dans ZooKeeper, que Lily HBase Indexer utilisera pour pointer vers la bonne collection dans Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumentation :
-n mdds_indexer– spécifie le nom de l'indexeur qui sera enregistré dans ZooKeeper-c indexer-config.xml– fichier de configuration qui spécifiera le comportement de l'indexeur-cp solr.zk=localhost:2181/solr– spécifie l'emplacement de la configuration ZooKeeper et Solr. Cela devrait être mis à jour avec l'emplacement spécifique à l'environnement de ZooKeeper.-cp solr.collection=mdds_collection– spécifie quelle collection mettre à jour. Rappelez-vous l'étape de configuration de Solr où nous avons créé collection1.
Le index-config.xml fichier est relativement simple dans ce cas ; tout ce qu'il fait est de spécifier à l'indexeur quelle table regarder, la classe qui sera utilisée comme mappeur (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ), et l'emplacement du fichier de configuration Morphline. Par défaut, mapping-type est défini sur ligne , auquel cas le document Solr devient la ligne complète. Param name="morphlineFile" spécifie l'emplacement du fichier de configuration Morphlines. L'emplacement peut être un chemin absolu de votre fichier Morphlines, mais puisque vous utilisez Cloudera Manager, spécifiez le chemin relatif comme morphlines.conf.
Le contenu du fichier de configuration hbase-indexer peut être trouvé ici.
Configuration et démarrage de Lily HBase Indexer
Lorsque vous activez Lily HBase Indexer, vous devez spécifier la logique de transformation Morphlines qui permettra à cet indexeur d'analyser les mises à jour de la table Medical Device et d'extraire tous les champs pertinents. Allez dans Services et choisissez Lily HBase Indexer que vous avez ajouté précédemment. Sélectionnez Configurations->Afficher et modifier->Service-Wide->Morphlines . Copiez et collez le fichier Morphlines.
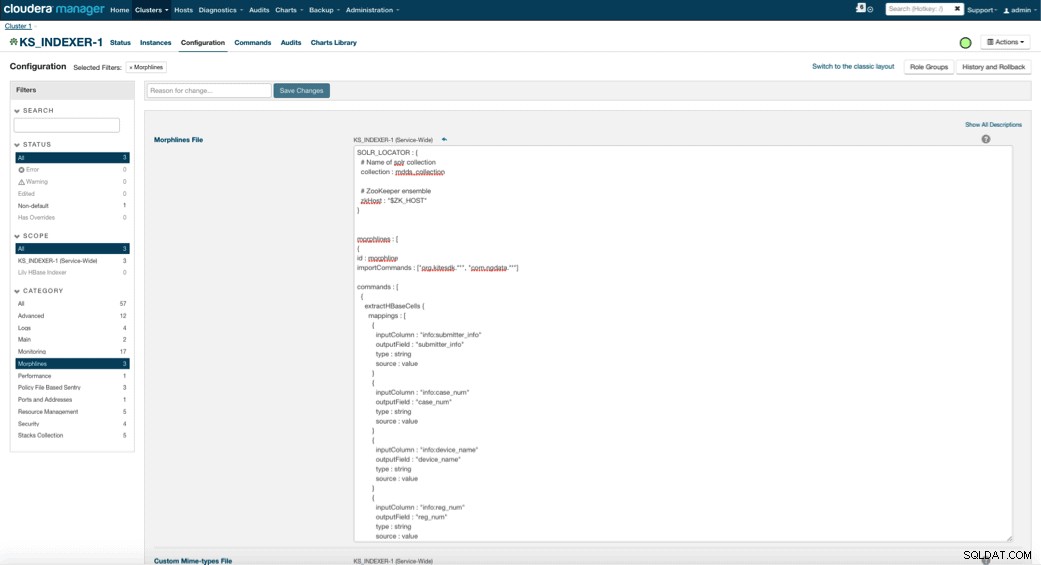
La bibliothèque de morphlines des dispositifs médicaux effectuera les actions suivantes :
- Lire les événements d'e-mail HBase avec le
extractHBaseCellscommande - Convertir la date/les horodatages dans un champ que Solr comprendra, avec le
convertTimestampcommandes - Supprimer tous les champs supplémentaires que nous n'avons pas spécifiés dans schema.xml, avec le
sanitizeUknownSolrFieldscommande
Téléchargez une copie de ce fichier Morphlines à partir d'ici.
Une remarque importante est que le champ id sera automatiquement généré par Lily HBase Indexer. Ce paramètre est configurable dans le fichier index-config.xml ci-dessus en spécifiant l'attribut unique-key-field. Il est recommandé de laisser le nom par défaut de l'id. Comme il n'a pas été spécifié dans le fichier xml ci-dessus, le champ d'id par défaut a été généré et sera une combinaison de RowID.
Accéder aux données
Vous avez le choix entre de nombreux outils visuels pour accéder aux images indexées. HUE et Solr GUI sont deux très bonnes options. HBase permet également un certain nombre de techniques d'accès, non seulement à partir d'une interface graphique, mais également via le shell HBase, l'API et même des techniques de script simples.
L'intégration avec Solr vous offre une grande flexibilité et peut également fournir des options de recherche très simples et avancées pour vos données. Par exemple, la configuration du fichier Solr schema.xml de sorte que tous les champs de l'objet e-mail soient stockés dans Solr permet aux utilisateurs d'accéder aux corps de message complets via une simple recherche, avec le compromis entre l'espace de stockage et la complexité de calcul. Alternativement, vous pouvez configurer Solr pour stocker uniquement un nombre limité de champs, tels que l'identifiant. Avec ces éléments, les utilisateurs peuvent rapidement rechercher Solr et récupérer le rowID qui à son tour peut être utilisé pour récupérer des champs individuels ou l'image entière à partir de HBase elle-même.
L'exemple ci-dessus ne stocke que le rowID dans Solr mais indexe tous les champs extraits de l'image. La recherche de Solr dans ce scénario récupère les ID de ligne HBase, que vous pouvez ensuite utiliser pour interroger HBase. Ce type de configuration est idéal pour Solr car il réduit les coûts de stockage et tire pleinement parti des capacités d'indexation de Solr.
Exemples de requêtes
Vous trouverez ci-dessous quelques exemples de requêtes pouvant être effectuées depuis l'application vers Solr. L'idée est que le client interrogera initialement les index Solr, renvoyant le rowID de HBase. Ensuite, interrogez HBase pour le reste des champs et/ou l'image brute d'origine.
- Donnez-moi tous les documents qui ont été déposés entre les dates suivantes :
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received :[2010-01 -06T23:59:59.999Z AU 2010-02-06T23:59:59.999Z]
- Donnez-moi les documents qui ont été classés sous le nom réglementaire des systèmes à rayons X mobiles :
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile système à rayons X
- Donnez-moi tous les documents déposés par les fabricants chinois :
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Les identifiants des documents Solr sont les identifiants de ligne dans HBase ; la deuxième partie de la requête sera à HBase pour extraire les données (y compris le PDF brut si nécessaire).
Accès via HUE
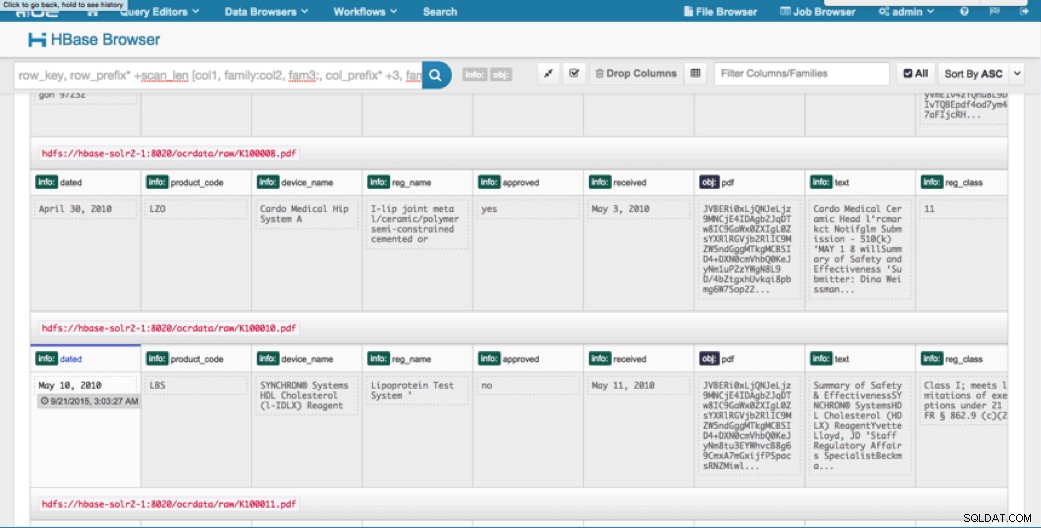
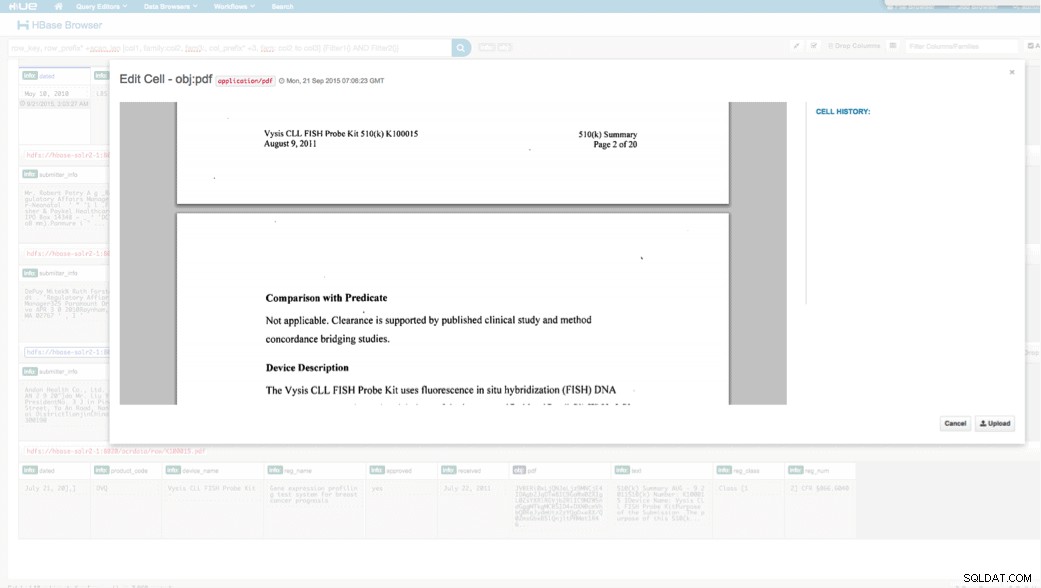
Nous pouvons afficher les données téléchargées via le navigateur HBase dans HUE. L'un des avantages de HUE est qu'il peut détecter les fichiers binaires pour PDF et les afficher lorsque vous cliquez dessus.
Vous trouverez ci-dessous un instantané de la vue des champs analysés dans les lignes HBase ainsi qu'une vue rendue de l'un des objets PDF stockés en tant que MOB dans la famille de colonnes obj.
Conclusion
Dans cet article, nous avons montré comment utiliser les technologies open source standard pour effectuer l'OCR sur des documents numérisés à l'aide d'un programme Spark évolutif, en les stockant dans HBase pour une récupération rapide et en indexant les informations extraites dans Solr. Il devrait être évident que :
- Étant donné le format de spécification du message, nous pouvons extraire des champs et des paires de valeurs et les rendre consultables via Solr.
- Ces champs de données peuvent répondre aux exigences de l'IDMP concernant la dématérialisation des anciennes données, qui entrera en vigueur l'année prochaine.
- Les champs ainsi que les images brutes peuvent être conservés dans HBase et accessibles via des API standard.
Si vous avez besoin de traiter des documents numérisés et de combiner les données avec diverses autres sources de votre entreprise, envisagez d'utiliser une combinaison de Spark, HBase, Solr, ainsi que Tesseract et Leptonica. Cela peut vous faire économiser beaucoup de temps et d'argent !
Jeff Shmain est architecte de solutions senior chez Cloudera. Il a plus de 16 ans d'expérience dans le secteur financier avec une solide compréhension du commerce des valeurs mobilières, des risques et des réglementations. Au cours des dernières années, il a travaillé sur diverses implémentations de cas d'utilisation dans 8 des 10 plus grandes banques d'investissement au monde.
Vartika Singh est consultante senior en solutions chez Cloudera. Elle a plus de 12 ans d'expérience dans l'apprentissage automatique appliqué et le développement de logiciels.